成都生物所開發(fā)一種從動物全基因組數(shù)據(jù)中提取并組裝線粒體全基因組的方法
作者:宋夢洹、李家堂
時間:2022-01-19
線粒體DNA因具有母系遺傳、跨階元保守以及較快的進化速率等特點,被廣泛應用于進化生物學,生物多樣性及臨床醫(yī)學的相關研究中。而線粒體基因組相比于線粒體DNA片段,包含更豐富的遺傳信息且不易受到自然選擇的影響,是一種重要的分子標記。
數(shù)年前,若要獲取一個完整的線粒體基因組,通常要使用噬菌體克隆的方法。在一代測序普及后,獲取完整線粒體基因組則通常需要使用引物步移(primer-walking)PCR的方法。但這兩種方法都非常耗時且成本較高。近年來,隨著測序技術的快速發(fā)展,動物(尤其是非模式動物)全基因組數(shù)據(jù)(WGS)獲取變得簡單、便宜;這使得數(shù)據(jù)庫中全基因組數(shù)據(jù)量呈指數(shù)態(tài)勢增長。全基因組測序數(shù)據(jù)往往同時包含線粒體序列和核基因序列,通過生物信息學的方法從中提取并組裝完整的線粒體基因組,是非常好的技術手段。雖然已有部分學者開發(fā)了一些生物信息學軟件用以實現(xiàn)此目的,但是,其大都具有自身的局限性,包括需要人工提供參考序列(“種子”)、數(shù)據(jù)兼容性差、準確率低等難點,未能高效、準確、便捷地完成相關任務。
為克服這一難點,提升研究人員的工作效率,中國科學院成都生物研究所李家堂課題組開發(fā)了一種不依賴“種子”序列從動物全基因組數(shù)據(jù)中提取并組裝線粒體全基因組的方法,并使用Python及C++編寫為軟件——MEANGS。MEANGS工作流程比較簡單,極大地提升了研究人員的可操作性。首先,利用預置的線粒體模塊數(shù)據(jù)庫,MEANGS使用nhmmer根據(jù)線粒體序列特征對輸入的二代數(shù)據(jù)進行預篩選并獲取潛在的線粒體編碼reads;獲得潛在的線粒體編碼reads后,MEANGS利用線性迭代算法(SSAKE)(C++改寫)組裝模塊對reads進行組裝獲取線粒體編碼contigs;相關contigs經(jīng)由nhmmer進行二次篩選,非冗余線粒體編碼contigs將被選擇作為“種子”序列用于全線粒體基因組的組裝;最后利用“種子”序列,MEANGS再次使用組裝模塊以“種子”contigs為基礎,組裝完整的線粒體基因組。另外,針對組裝好的線粒體基因組,MEANGS可對編碼基因進行輔助注釋。

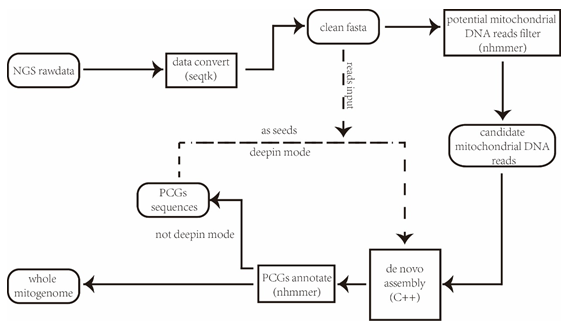
圖1 MEANGS利用動物全基因組數(shù)據(jù)組裝線粒體基因組的工作流程。
針對MEANGS,本研究使用了來自不同類群(哺乳類、鳥類、魚類、爬行類、兩棲類,昆蟲和軟體動物),不同測序質(zhì)量的16組數(shù)據(jù)來進行相關測試。同時,本研究選取了其他三款同類型軟件(norgal, NOVOPlasty, MitoZ)進行同條件測試,并基于常規(guī)的評價標準(運行時長、內(nèi)存使用、準確性、完整性、完成數(shù)量等)對軟件的表現(xiàn)進行評價。測試及比較結(jié)果表明,MEANGS數(shù)據(jù)兼容性最好,完成了所有測試數(shù)據(jù)的測試,而其他三款軟件均有失敗案例。在常規(guī)的評價指標上,MEANGS表現(xiàn)出最優(yōu)的整體性能,除了在內(nèi)存使用上稍遜色于norgal,其余表現(xiàn)均優(yōu)于其他軟件。MEANGS的開發(fā)有利于研究人員開展相關工作,推動相關學科的高質(zhì)量快速發(fā)展。
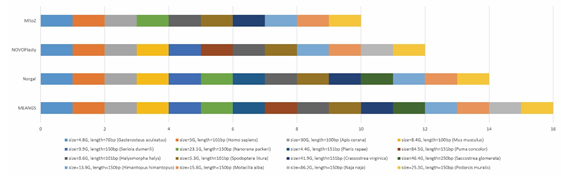
圖2 MEANGS與其他三款軟件相比表現(xiàn)出最好的數(shù)據(jù)兼容性。
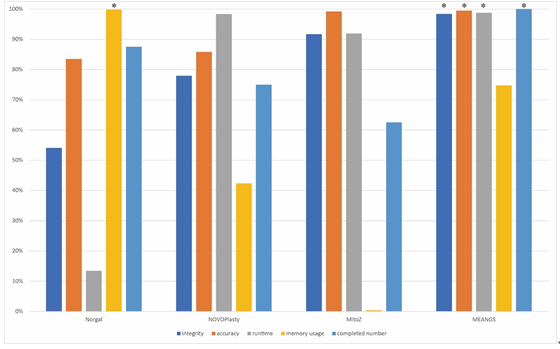
圖3 MEANGS與其他三款軟件在常規(guī)評價指標上顯示出明顯優(yōu)勢。
該研究成果以MEANGS: an efficient seed-free tool for de novo assembling animal mitochondrial genome using whole genome NGS data為題,已在生物信息學國際頂級期刊Briefings in Bioinformatics(IF: 11.62)在線發(fā)表。中國科學院成都生物研究所在讀博士研究生宋夢洹,特別研究助理嚴超超博士為論文的共同第一作者,李家堂研究員為論文的通訊作者。本研究得到成都生物研究所高級工程師蔣海波博士和碩士生桂承波的大力幫助。本研究得到中國科學院B類先導科技專項、科技部第二次青藏高原綜合科學考察研究項目、中國科學院對外合作重點項目、中國科學院西部之光交叉團隊項目、中國科學院前沿科學重點研究項目及四川省杰出青年科技人才項目等項目的資助。




