成都生物研究所在沙蜥屬蜥蜴線粒體基因組重排研究獲進展
作者:吳娜、郭憲光
時間:2022-02-28
動物線粒體基因由于其母系遺傳、易擴增及進化速率快等特點,被廣泛用于分子系統發育、種群遺傳、譜系地理和歷史生物地理等研究領域。與此同時,有關線粒體基因組的結構特征與進化一直是進化生物學的研究熱點。眾所周知,脊椎動物的線粒體基因組的組成和結構相對保守,而在兩棲爬行動物中發現了大量的基因重排、基因重復和丟失的現象。然而,種內水平的基因重排較為罕見。
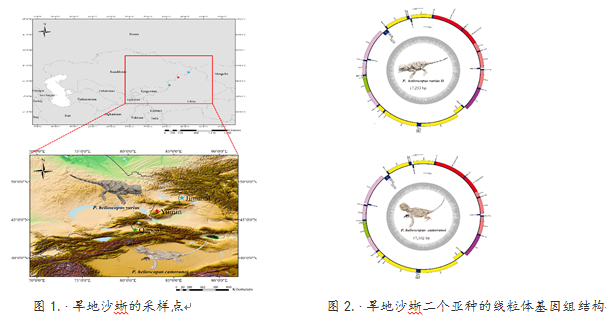
近日,中科院成都生物所郭憲光團隊采用二代測序技術,獲得了采自新疆的旱地沙蜥(Phrynocephalus helioscopus)兩個亞種(準噶爾亞種P. helioscopus varius和伊犁亞種P. helioscopus cameranoi)的線粒體全基因組(圖1、圖2)。
結合GenBank中沙蜥屬物種的線粒體全基因組(共15個物種、25條)進行全面的比較分析,梳理該屬的線粒體基因組特征與進化。通過基因排列順序和系統發育分析,發現沙蜥屬中有六種基因重排類型,且有較為普遍的種內基因重排現象,比如在旱地沙蜥、紅尾沙蜥P. erythrurus、青海沙蜥P. vlangalii和南疆沙蜥P. forsythii均有發現(圖3)。總體而言,基因重排的發生可能是由多個獨立的結構動力事件引起的。進而發現,旱地沙蜥的這兩個亞種有23個tRNA,有tRNA-Phe基因重復,基于串聯重復/隨機丟失(TDRL)模型可推測其基因重排機制(圖4)。運用松弛分子鐘估算分析,表明旱地沙蜥的這兩個亞種大約在234萬年前(95% CI: 135--373萬年前)發生分化。當基因復制后,大約在72--23萬年前發生了替代丟失,這暗示了這些重排基因的重復與固定可以相當快地發生,而基因重排的發生可能是多個獨立的事件共同作用的結果。
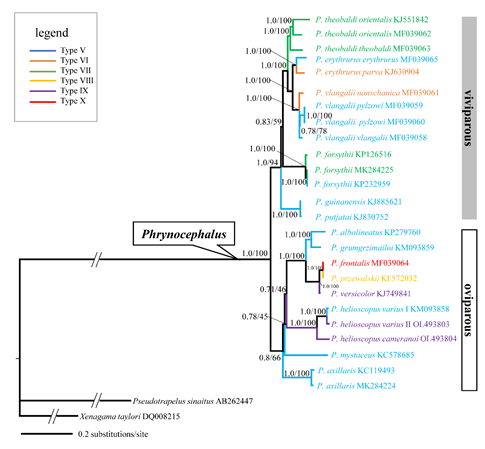
圖3. 基于串聯PCGs基因構建的系統發育樹
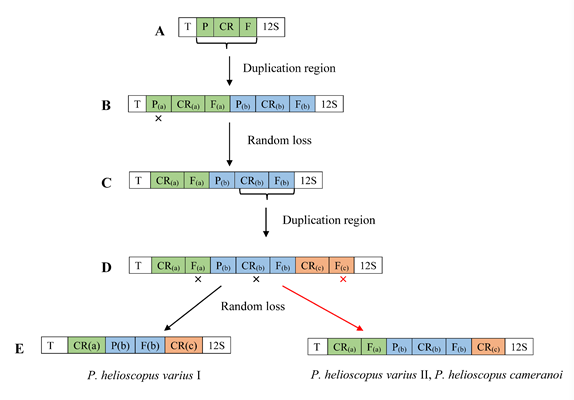
圖4. 基于串聯重復/隨機丟失(TDRL)模型推測基因重排過程
相關研究成果以“Comparative Analysis of Mitochondrial Genomes in Two Subspecies of the Sunwatcher Toad-Headed Agama (Phrynocephalus helioscopus): Prevalent Intraspecific Gene Rearrangements in Phrynocephalus”為題發表在國際期刊Genes(IF: 4.096)。本研究提示,需要在種群水平上獲得更多的線粒體基因組數據,從而揭示沙蜥屬中普遍的種內線粒體基因組重排式樣及其機制。中科院成都生物所在讀碩士生吳娜為該論文的第一作者,郭憲光副研究員為通訊作者。本研究得到中科院“絲路環境”先導專項(XDA20050201)、第三次新疆綜合科學考察項目(2021xjkk0600)和國家自然科學基金項目(32070433, 32000288)的資助。




